- Кэп
-
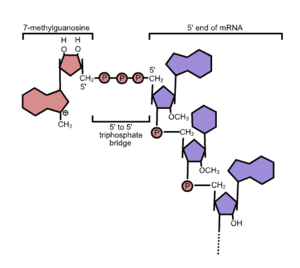
Кэп (5'-кэп, кэп-структура) (от англ. cap — шапочка) — один или несколько модифицированных нуклеотидов на 5'-конце транскриптов, синтезированных РНК-полимеразой II. С химической точки зрения, кэп представляет собой 7-метилгуанозин, соединённый 5',5'-трифосфатным мостиком с первым нуклеотидным остатком транскрипта. В узком смысле под кэпом понимают именно 7-метилгуанозин. Кроме того, первые два нуклеотида транскрипта могут метилироваться по 2'-O-положению рибозы. Кэп способствует эффективному процессингу пре-мРНК, экспорту мРНК из ядра, её трансляции и защите от быстрой деградации[1][2].
Содержание
Типы кэп-структуры и их распространённость
У подавляющего большинства эукариот 5'-конец транскриптов, синтезируемых РНК-полимеразой II, ко-транскрипционно модифицируется путём присоединения 7-метилгуанозина, или кэпа (см. рисунок). РНК-полимераза II синтезирует все пре-мРНК, некоторые малые ядерные РНК и малые ядрышковые РНК. Вирусы, которые приспособлены к жизни в эукариотических клетках, также могут иметь кэпированные РНК независимо от того, синтезируются ли они РНК-полимеразой II или другим ферментом[3]. В зависимости от типа организма и типа РНК первоначальная кэп-структура может подвергаться дальнейшим модификациям, главным образом, метилированию[4].
Выделяют следующие типы кэпа:
- кэп 0 (m7GpppNp, где N — любой нуклеотид) — это минимальная кэпирующая структура, которая представляет собой 7-метилгуанозин, соединённый 5',5'-трифосфатным мостиком с первым нуклеотидом РНК[2]. Кэп 0 распознаётся фактором инициации трансляции eIF4E[5];
- кэп 1 (m7GpppNm2'-Op) отличается от кэпа 0 метилированием первого нуклеотида транскрипта по 2'-O положению рибозы;
- кэп 2 (m7GpppNm2'-OpNm2'-Op) отличается от кэпа 0 метилированием первого и второго нуклеотидов транскрипта по 2'-O положению рибозы;
- кэп 4 (m7GpppNm2'-OpNm2'-OpNm2'-OpNm2'-Op);
- 2,2,7-триметилгуанозиновый кэп (m2,2,7GpppNp)
Кэп 0 характерен для РНК растений (а также вирусов растений) и грибов, у животных он встречается редко. Для РНК животных характерны кэп 1 и кэп 2, соотношение которых в клетке зависит от вида организма. Вирусы животных также, как правило используют кэп 1 или кэп 2. Интересно, что в РНК вирусов животных первым нуклеотидом после 7-метилгуанозина обычно является пурин, который может быть дополнительно метилирован по атомам азотистого основания (например, с образованием N6-метиладенозина). В клеточных же мРНК первым нуклеотидом после кэпа может быть любой из четырёх, и он тоже, как правило, дополнительно метилирован. В целом можно сказать, что чем более высоко организован организм, тем больше метильных групп содержится в его кэп-структурах[2]. Кэп 4 был обнаружен у некоторых простейших[6]. 2,2,7-триметилгуанозиновый кэп характерен для малых ядерных РНК и служит сигналом к их транспорту и/или удержанию в ядре[4].
Механизм кэпирования
Кэпирующие ферменты
 Строение молекулы рибозы, показывающее положение 2'-, 3'- и 5'- атомов углерода
Строение молекулы рибозы, показывающее положение 2'-, 3'- и 5'- атомов углерода
Кэпирование — первый этап процессинга РНК. Кэпирование происходит ко-траскрипционно в ядре, когда синтезируемый транскрипт достигает длины 25—30 нуклеотидов[7]. Кэпирование осуществляется тремя ферментами: РНК-трифосфатазой, гуанилтрансферазой и 7-метилтрансферазой[8].
РНК-трифосфатаза отщепляет γ-фосфатную группу от 5'-концевого нуклеотида транскрипта. Аминокислотная последовательность РНК-трифосфатаз существенно варьирует среди эукариот, и можно выделить по крайней мере два семейства данных ферментов: РНК-трифосфатазы, зависимые от бивалентных катионов (характерны для простейших, грибов и вирусов эукариот), и РНК-трифосфатазы, независимые от бивалентных катионов (характерны для животных и растений)[9]. У дрожжей Saccharomyces cerevisiae РНК-трифосфатаза кодируется собственным геном Cet1, в то время как у млекопитающих синтезируется бифункциональный фермент, который обладает и РНК-трифосфатазной (N-концевой домен), и гуанилтрансферазной активностью (C-концевой домен). Гуанилтрансфераза (у дрожжей кодируется геном Ceg1) осуществляет перенос остатка ГМФ из ГТФ на β-фосфатную группу 5'-концевого нуклеотида транскрипта с формированием структуры GpppN. Эта реакция происходит в два этапа с образованием промежуточного соединения фермент-(лизил-N)-ГМФ. 7-метилтрансфераза у всех организмов кодируется отдельным геном (Abd1 у дрожжей)[9]. Этот фермент катализирует перенос метильной группы от S-аденозилметионина на Gppp-РНК с образованием m7Gppp-РНК.
Связь кэпирования с транскрипцией
Ко-транскрипционное протекание процесса кэпирования обеспечивается тем, что кэпирующие ферменты прямо связываются с фосфорилированным C-концевым доменом большой субъединицы РНК-полимеразы II[10][11]. С-концевой домен имеет уникальную эволюционно консервативную структуру: он состоит из многократно повторяющихся аминокислотных мотивов Tyr-Ser-Pro-Thr-Ser-Pro-Ser[9]. Несколько киназ могут фосфорилировать аминокислотные остатки в C-концевом домене. Переход от инициации транскрипции к ранней элонгации сопровождается фосфорилированием Ser-5 фактором транскрипции TFIIH[12]. В ходе транскрипции количество фосфорилированного Ser-5 снижается, и начинает преобладать фосфорилированный Ser-2[9]. После фосфорилирования РНК-полимеразы II по Ser-5 к транскрипционному комплексу присоединяется негативный транскрипционный фактор DSIF (англ. DRB sensitivity inducing factor), который в свою очередь привлекает второй негативный регулятор — NELF (англ. negative elongation factor). Вместе эти факторы временно ингибируют дальнейшее прохождение транскрипции. Гуанилтрансферазный домен кэпирующего фермента млекопитающих имеет сродство к С-концевому домену РНК-полимеразы II, фосфорилированному по Ser-5. У дрожжей с РНК-полимеразой связывается гуанилтрансфераза Ceg1, которая затем привлекает в комплекс РНК-трифосфатазу Cet1. Кроме того, гуанилтрансфераза связывается одновременно и с одной из субъединиц фактора DSIF. Связывание с фосфорилированной формой РНК-полимеразы и с DSIF стимулирует каталитическую активность гуанилтрансферазы[13]. Описанные взаимодействия обеспечивают прохождение кэпирования вскоре после инициации транскрипции и до того, как транскрипционный комплекс перейдёт к продуктивной элонгации. Считается, что фосфорилированный Ser-5 пропадает к тому моменту, как транскрипт достигает длины 500 нуклеотидов. К этому же времени из транскрипционного комплекса диссоциируют и кэпирующие ферменты[12]. Важно отметить, что не только транскрипция определяет ход кэпирования, но и успешность процесса кэпирования оказывает влияние на дальнейший ход транскрипции (см. ниже).
Функции
Кэпирование 5'-конца РНК-транскрипта во многом определяет его дальнейшую судьбу в клетке. Известны следующие функции кэпа:
- регуляция транскрипции;
- участие в сплайсинге;
- участие в процессинге 3'-конца мРНК;
- регуляция транспорта РНК между ядром и цитоплазмой;
- защита транскрипта от деградации под действием экзонуклеаз;
- стимуляция трансляции.
Регуляция транскрипции
Ряд исследований указывает на то, что кэпирующие ферменты могут играть роль в регуляции транскрипции[13]. В 2007 году было сделано важное открытие — оказалось, что промоторы очень многих генов эукариотических организмов постоянно содержат полностью собранный инициаторный комплекс, который может синтезировать короткие транскрипты, однако дальнейшее продвижение этого комплекса внутрь гена заингибировано[14][15][16]. Предполагают, что такая система позволяет при необходимости быстро начать транскрипцию, что особенно важно в случае генов, отвечающих за эмбриональное развитие и ответ клетки на внешние воздействия. Механизм переключения транскрипции с «паузы» на продуктивную элонгацию сейчас рассматривается как ключевой способ контроля за транскрипцией. Есть основания полагать, что кэпирование может быть одним из таких «переключателей»[17].
Считается, что движение транскрипционного комплекса ингибируется факторами транскрипции DSIF и NELF, которые действуют в паре, и восстанавливается под действием положительного регулятора PTEFb, который фосфорилирует повторяющиеся аминокислотные мотивы в С-концевом домене РНК-полимеразы II по положению Ser-2[13]. Несколькими группами исследователей было установлено, что транскрипция генов у дрожжей стимулируется кэпирующими ферментами[18][19][20]. При этом выяснилось, что действие этих ферментов на транскрипцию не изменяется, даже если они оказываются каталитически неактивными вследствие мутаций. Этот факт позволяет предположить, что само по себе присутствие кэпирующих ферментов в транскрипционном комплексе, а не обязательно даже кэп-структура, стимулирует движение транскрипционного комплекса. Стимулирующее действие кэпирующих ферментов на транскрипцию можно объяснить, во-первых, тем что они способны связываться с DSIF, вытесняя NELF из комплекса с ним, в результате чего ингибиторное действие DSIF на транскрипцию прекращается[19]. Во-вторых, 7-метилтрансфераза (по крайней мере, в случае Schizosaccharomyces pombe и Caenorhabditis elegans) может привлекать в транскрипционный комплекс фактор транскрипции PTEFb, что способствует началу продвижения комплекса внутрь гена[21][22]. Кроме того, дополнительное привлечение PTEFb обеспечивается кэп-связывающим белковым комплексом (CBC) (см. ниже)[23].
В то же время есть данные, что транскрипция, по крайней мере, некоторых генов дрожжей происходит независимо от кэпирования[13]. То есть, по крайней мере, у дрожжей связь кэпирования и транскрипции является геноспецифической характеристикой. Дальнейшие исследования должны показать, так ли это в случае других организмов.
Участие в сплайсинге
Кэп-структура стимулирует сплайсинг пре-мРНК как in vitro, так и in vivo, причём в большей степени стимулируется вырезание интрона ближайшего к 5'-концу транскрипта[24][25][26]. Позитивное действие кэпа на сплайсинг объясняется следующим образом. Сразу после присоединения кэпа к 5'-концу транскрипта с ним связывается кэп-связывающий комплекс CBC (англ. cap binding complex), который важен для последующих этапов процессинга пре-мРНК. CBC состоит из двух субъединиц: кэп-связывающей CBP20 (англ. cap binding protein) и вспомогательной CBP80[27]. Кэп-связывающий комплекс взаимодействует с одним из компонентов сплайсосомы, мяРНП U1, и обеспечивает его посадку на пре-мРНК недалеко от 5'-конца. мяРНП U1 отвечает за распознавание 5'-концевого сайта сплайсинга, с него начинается последующая сборка сплайсосомы[28]. Стоит отметить, что как и в случае с транскрипцией, пока что точно не известно, какова доля пре-мРНК, сплайсинг которых не зависит от наличия кэпа, у разных организмов. Во всяком случае, показано, что такая зависимость является геноспецифической у S. cerevisiae[13].
Участие в процессинге 3'-конца мРНК
3'-конец мРНК эукариот формируется в два этапа: сначала особая эндонуклеаза вносит разрыв в 3'-концевой участок мРНК, а затем поли(А)-полимераза присоединяет ко вновь сформированному 3'-концу поли(А)-хвост[29]. Наличие кэпа стимулирует эндопротеолитическое расщепление 3'-конца мРНК в ядерных экстрактах клеток HeLa[30][31]. Положительное действие кэпа в этом случае также осуществляется через кэп-связывающий комплекс, который связывается с компонентами 3'-процессирующего комплекса и обеспечивает его стабильность[32]. Влияет ли наличие кэпа на эффективность полиаденилирования пока точно не установлено.
Роль в транспорте РНК
Кэп играет важную роль транспорте РНК из ядра. Экспорт мРНК осуществляется при участии комплекса транспортных факторов Mex67—Mtr2 (у дрожжей) или TAP—p15 (у многоклеточных)[33]. Однако этот комплекс связывается с мРНК не напрямую, а через адаптерный белок Yra1 (у дрожжей) или ALY/REF (у многоклеточных), который является одной из субъединиц белкового комплекса TREX. В свою очередь, TREX привлекается в комплекс с мРНК за счёт прямого взаимодействия ALY/REF с CBC80 субъединицей кэп-связывающего комплекса[34]. Такой механизм обеспечивает присоединение транспортного комплекса близко к 5'-концу мРНК и соответствующую направленность её транспорта, 5'-концом в сторону цитоплазмы.
Малые ядерные РНК, синтезированные РНК-полимеразой II, экспортируются в цитоплазму на некоторое время для дальнейшего созревания, после чего возвращаются обратно в ядро для выполнения своих функций, при этом кэп регулирует их транспорт в обоих направлениях. мяРНК экспортируются при участии транспортного белка Crm1, который, как и в случае с мРНК, связывается со своим субстратом через адаптерный белок PHAX (англ. phosphorylated adaptor for RNA export)[35]. PHAX присоединяется к мяРНК благодаря сродству к кэп-связывающему комплексу. В ходе формирования мяРНП в цитоплазме кэп-структура мяРНК подвергается двойному метилированию с образованием 2,2,7-триметилгуанозинового кэпа[4]. Другой транспортный фактор, снурпортин 1, узнаёт такой модифицированный кэп и обеспечивает транспорт мяРНП обратно в ядро[36]. Вероятно, что дополнительное метилирование кэпа также предотвращает повторный или случайный экспорт РНК из ядра и/или их возвращение в ядро после митоза.
Защита мРНК от деградации
Присутствие кэпа на 5'-конце защищает молекулы мРНК от быстрой деградации под действием экзонуклеаз двумя способами[37][2]. Во-первых, 5'-экзонуклеазы не могут расщеплять 5',5'-трифосфатную связь между кэпом и телом мРНК. Во-вторых, кэп-связывающие белки (например, eIF4E—eIF4G) блокируют доступ нуклеаз к 5'-концу мРНК[4]. Отщепление кэпа (декэпирование) является одной из ключевых стадий в некоторых путях деградации мРНК (см. ниже).
Роль в трансляции
После завершения процессинга мРНК следует своеобразный этап проверки его качества. Благодаря процессу расщепления мРНК, содержащих преждевременные стоп-кодоны (англ. Nonsense-mediated decay (NMD)) клетка избавляется от мРНК, на которых могут синтезироваться укороченные и вероятно неспособные выполнять свои функции белки. Зрелая мРНК, которая по-прежнему ещё содержит кэп-связывающий комплекс CBC на 5'-конце и другие белки, характерные для ядерных мРНП, вовлекается в первый пробный раунд трансляции. Если в ходе трансляции обнаруживается присутствие преждевременного стоп-кодона, то такая мРНК подвергается деградации по пути NMD. Если же такого стоп-кодона не было, то происходит замена мРНК-связывающих белков на характерные для цитоплазмы (например, PABP2 заменяется на PABP1, CBC — на eIF4E), и мРНК становится полноценной матрицей для трансляции[38].
Большая часть мРНК эукариот транслируется по кэп-зависимому механизму и только относительно небольшая их доля — по механизму внутренней посадки рибосомы. Относительно давно уже было известно, что некэпированные мРНК являются плохими матрицами для синтеза белка в экспериментах in vitro и что наличие кэпа стимулирует связывание мРНК с рибосомой[2]. На сегодняшний день известен молекулярный механизм такого действия кэпа. Инициация кэп-зависимой трансляции включает этап сборки комплекса eIF4F (eIF4E—eIF4G—eIF4A) на кэпированном 5'-конце мРНК. Первым к мРНК присоединяется кэп-связывающий фактор инициации трансляции eIF4E, который привлекает в комплекс более крупный белок eIF4G. eIF4G, в свою очередь, служит платформой для посадки других белков: eIF4A, eIF3 и PABP. Действие этих и ещё некоторых других белков подготавливает мРНК для посадки 43S преинициаторного комплекса, содержащего малую субъединицу рибосомы. После этого следует сканирование малой субъединицей рибосомы 5'-нетранслируемой области мРНК, начиная с 5'-конца, в поисках стартового кодона и начало синтеза белка[39][40].
Декэпирование и рекэпирование
Декэпирование
Кэп наряду с поли(А)-хвостом обеспечивает стабильность молекулы мРНК, а отщепление кэпа ведёт к её деградации. Таким образом, декэпирование является критическим моментом в жизненном цикле мРНК и строго регулируется в клетке[41].
Известно несколько возможных путей деградации эукариотической мРНК[42]:
- деградация, зависимая от укорочения поли(А)-хвоста;
- 5'→3'-деградация;
- 3'→5'-деградация;
- деградация, не зависимая от укорочения поли(А)-хвоста;
- деградация при участи эндонуклеаз.
В случае зависимой от укорочения поли(А)-хвоста деградации мРНК события могут развиваться по двум не исключающим друг друга сценариям. Расщепление в направлении 5'→3' начинается с декэпирования мРНК под действием декэпирующего белкового комплкекса. У S. cerevisiae этот комплекс состоит из двух белков: каталитической субъединицы Dcp2 (англ.)русск. и ко-активатора Dcp1 (англ.)русск.. У высших эукариот в этот комплекс входит третий белок, называемый Hedls (у человека), который обеспечивает дополнительную связь между субъединицами комплекса и стимулирует декэпирование[41]. Продуктами реакции декэпирования являются 7-метил-ГДФ и РНК с монофосфатом на 5'-конце. Такой 5'-конец становиться доступным экзорибонуклеазе Xrn1 (англ.)русск., которая разрушает мРНК в направлении 5'→3'. Белки, участвующие в 5'→3'-деградации мРНК, обнаруживаются в большом количестве в тельцах процессинга (англ.)русск., которые рассматриваются как возможное место хранения и/или деградации мРНК[42].
Разрушение мРНК в направлении 3'→5' катализируется крупной мультисубъединичной экзонуклеазой — экзосомой (англ.)русск.. Кэпированный ди- или олигонуклеотид, который остаётся после завершения работы экзосомы, подвергается декэпированию под действием фермента DcpS (англ.)русск. с образованием 7-метил-ГМФ. DcpS также превращает 7-метил-ГДФ, который образуется при декэпировании мРНК под действием декэпирующего комплекса, в 7-метил-ГМФ[4].
Рекэпирование
Существуют данные о том, что декэпированные мРНК могут повторно кэпироваться в цитоплазме[1]. В 2009 году было подтверждено более раннее предположение о том, что укороченные мРНК, которые образуются в результате неполного расщепления по пути NMD, подвергаются 5'-концевой модификации неотличимой от кэпирования. Кроме того, были обнаружены цитоплазматические ферменты, формирующие единый комплекс и обеспечивающие последовательное присоединение β-фосфата и ГМФ к монофосфату на 5'-конце укороченной РНК. В результате этих двух реакций происходит формирование структуры GpppN[43]. Хотя 7-метилтрансфераза присутствует в цитоплазме, она не входит в состав цитоплазматического кэпирующего комплекса. Каким именно происходит метилирование кэпа при цитоплазматическом кэпировании ещё предстоит установить[1]. Также до сих пор не известно, какое биологическое значение может иметь рекэпирование.
Примечания
- ↑ 1 2 3 Schoenberg D. R., Maquat L. E. (2009). «Re-capping the message». Trends Biochem Sci. 34: 435—442. PMID 19729311.
- ↑ 1 2 3 4 5 Banerjee A. K. (1980). «5'-terminal cap structure in eucaryotic messenger ribonucleic acids». Microbiol Rev. 44: 175—205. PMID 6247631.
- ↑ Decroly E, Ferron F, Lescar J, Canard B. (2011). «Conventional and unconventional mechanisms for capping viral mRNA.». Nat Rev Microbiol. 10: 51—65. DOI:10.1038/nrmicro2675. PMID 22138959.
- ↑ 1 2 3 4 5 Cougot N., van Dijk E., Babajko S., Séraphin B. (2004). «'Cap-tabolism'.». Trends Biochem Sci. 29: 436—444. PMID 15362228.
- ↑ Marcotrigiano J., Gingras A. C., Sonenberg N., Burley S. K. (1997). «Cocrystal structure of the messenger RNA 5' cap-binding protein (eIF4E) bound to 7-methyl-GDP.». Cell. 89: 951—961. DOI:10.1016/S0092-8674(00)80280-9. PMID 9200613.
- ↑ Perry K. L., Watkins K. P., Agabian N. (1987). «Trypanosome mRNAs have unusual "cap 4" structures acquired by addition of a spliced leader». Proc Natl Acad Sci U S A. 84: 8190—8194. PMID 3120186.
- ↑ Rasmussen E. B., Lis J. T. (1993). «In vivo transcriptional pausing and cap formation on three Drosophila heat shock genes». Proc Natl Acad Sci U S A. 90: 7923—7927. PMID 8367444.
- ↑ Shuman S. (2001). «Structure, mechanism, and evolution of the mRNA capping apparatus». Prog Nucleic Acid Res Mol Biol. 66: 1—40. PMID 11051760.
- ↑ 1 2 3 4 Gu M., Lima C. D. (2005). «Processing the message: structural insights into capping and decapping mRNA». Curr Opin Struct Biol. 15: 99—106. PMID 15718140.
- ↑ Cho E. J., Takagi T., Moore C. R., Buratowski S. (1997). «mRNA capping enzyme is recruited to the transcription complex by phosphorylation of the RNA polymerase II carboxy-terminal domain». Genes Dev. 11: 3319—3326. DOI:10.1101/gad.11.24.3319. PMID 9407025.
- ↑ McCracken S., Fong N., Rosonina E., Yankulov K., Brothers G., Siderovski D., Hessel A., Foster S., Shuman S., Bentley D. L. (1997). «5'-Capping enzymes are targeted to pre-mRNA by binding to the phosphorylated carboxy-terminal domain of RNA polymerase II». Genes Dev. 11: 3306—3318. DOI:10.1101/gad.11.24.3306. PMID 9407024.
- ↑ 1 2 Hocine S., Singer R. H., Grünwald D. (2010). «RNA processing and export». Cold Spring Harb Perspect Biol. 2: a000752. DOI:10.1101/cshperspect.a000752. PMID 20961978.
- ↑ 1 2 3 4 5 Cowling V. H. (2010). «Regulation of mRNA cap methylation». Biochem. J. 425 (2): 295—302. DOI:10.1042/BJ20091352. PMID 20025612.
- ↑ Guenther M. G., Levine S. S., Boyer L. A., Jaenisch R., Young R. A. (2007). «A chromatin landmark and transcription initiation at most promoters in human cells». Cell 130 (1): 77—88. DOI:10.1016/j.cell.2007.05.042. PMID 17632057.
- ↑ Muse G. W., Gilchrist D. A., Nechaev S., Shah R., Parker J. S., Grissom S. F., Zeitlinger J., Adelman K. (2007). «RNA polymerase is poised for activation across the genome». Nat. Genet. 39 (12): 1507—1511. DOI:10.1038/ng.2007.21. PMID 17994021.
- ↑ Zeitlinger J., Stark A., Kellis M., Hong J. W., Nechaev S., Adelman K., Levine M., Young R. A. (2007). «RNA polymerase stalling at developmental control genes in the Drosophila melanogaster embryo». Nat. Genet. 39 (12): 1512—1516. DOI:10.1038/ng.2007.26. PMID 17994019.
- ↑ Moore M. J., Proudfoot N. J. (2009). «Pre-mRNA processing reaches back to transcription and ahead to translation». Cell 136 (4): 688—700. DOI:10.1016/j.cell.2009.02.001. PMID 19239889.
- ↑ Kim H. J., Jeong S. H., Heo J. H., Jeong S. J., Kim S. T., Youn H. D., Han J. W., Lee H. W., Cho E. J. (2004). «mRNA capping enzyme activity is coupled to an early transcription elongation». Mol. Cell. Biol. 24 (14): 6184—6193. DOI:10.1128/MCB.24.14.6184-6193.2004. PMID 15226422.
- ↑ 1 2 Mandal S. S., Chu C., Wada T., Handa H., Shatkin A. J., Reinberg D. (2004). «Functional interactions of RNA-capping enzyme with factors that positively and negatively regulate promoter escape by RNA polymerase II». Proc. Natl. Acad. Sci. U.S.A. 101 (20): 7572—7577. DOI:10.1073/pnas.0401493101. PMID 15136722.
- ↑ Schroeder S. C., Zorio D. A., Schwer B., Shuman S., Bentley D. (2004). «A function of yeast mRNA cap methyltransferase, Abd1, in transcription by RNA polymerase II». Mol. Cell 13 (3): 377—387. DOI:doi:10.1016/S1097-2765(04)00007-3. PMID 14967145.
- ↑ Guiguen A., Soutourina J., Dewez M., Tafforeau L., Dieu M., Raes M., Vandenhaute J., Werner M., Hermand D. (2007). «Recruitment of P-TEFb (Cdk9-Pch1) to chromatin by the cap-methyl transferase Pcm1 in fission yeast». EMBO J. 26 (6): 1552—1559. DOI:10.1038/sj.emboj.7601627. PMID 17332744.
- ↑ Takagi T., Walker A. K., Sawa C., Diehn F., Takase Y., Blackwell T. K., Buratowski S. (2003). «The Caenorhabditis elegans mRNA 5'-capping enzyme. In vitro and in vivo characterization». J. Biol. Chem. 278 (16): 14174—14184. DOI:10.1074/jbc.M212101200. PMID 12576476.
- ↑ Lenasi T., Peterlin B. M., Barboric M. (2011). «Cap-binding protein complex links pre-mRNA capping to transcription elongation and alternative splicing through positive transcription elongation factor b (P-TEFb)». J. Biol. Chem. 286 (26): 22758—22768. DOI:10.1074/jbc.M111.235077. PMID 21536667.
- ↑ Konarska M. M., Padgett R. A., Sharp P. A. (1984). «Recognition of cap structure in splicing in vitro of mRNA precursors». Cell 38 (3): 731—736. DOI:10.1016/0092-8674(84)90268-X. PMID 6567484.
- ↑ Edery I., Sonenberg N. (1985). «Cap-dependent RNA splicing in a HeLa nuclear extract». Proc. Natl. Acad. Sci. U.S.A. 82 (22): 7590—7594. PMID 3865180.
- ↑ Ohno M., Sakamoto H., Shimura Y. (1987). «Preferential excision of the 5' proximal intron from mRNA precursors with two introns as mediated by the cap structure». Proc. Natl. Acad. Sci. U.S.A. 84 (15): 5187—5191. PMID 2440046.
- ↑ Topisirovic I., Svitkin Y. V., Sonenberg N., Shatkin A. J. (2011). «Cap and cap-binding proteins in the control of gene expression». Wiley Interdiscip Rev RNA 2 (2): 277—298. DOI:10.1002/wrna.52. PMID 21957010.
- ↑ Lewis J. D., Izaurralde E., Jarmolowski A., McGuigan C., Mattaj I. W. (1996). «A nuclear cap-binding complex facilitates association of U1 snRNP with the cap-proximal 5' splice site». Genes Dev. 10 (13): 1683—1698. DOI:10.1101/gad.10.13.1683. PMID 8682298.
- ↑ Lewis J. D., Izaurralde E. (1997). «The role of the cap structure in RNA processing and nuclear export». Eur. J. Biochem. 247 (2): 461—469. DOI:10.1111/j.1432-1033.1997.00461.x. PMID 9266685.
- ↑ Hart R. P., McDevitt M. A., Nevins J. R. (1985). «Poly(A) site cleavage in a HeLa nuclear extract is dependent on downstream sequences». Cell 43 (3 Pt 2): 677—683. DOI:10.1016/0092-8674(85)90240-5. PMID 2866847.
- ↑ Cooke C., Alwine J. C. (1996). «The cap and the 3' splice site similarly affect polyadenylation efficiency». Mol. Cell. Biol. 16 (6): 2579—2584. PMID 8649365.
- ↑ Flaherty S. M., Fortes P., Izaurralde E., Mattaj I. W., Gilmartin G. M. (1997). «Participation of the nuclear cap binding complex in pre-mRNA 3' processing». Proc. Natl. Acad. Sci. U.S.A. 94 (22): 11893—11898. PMID 9342333.
- ↑ Köhler A., Hurt E. (2007). «Exporting RNA from the nucleus to the cytoplasm». Nat. Rev. Mol. Cell Biol. 8 (10): 761—773. DOI:10.1038/nrm2255. PMID 17786152.
- ↑ Cheng H., Dufu K., Lee C. S., Hsu J. L., Dias A., Reed R. (2006). «Human mRNA export machinery recruited to the 5' end of mRNA». Cell 127 (7): 1389—1400. DOI:10.1016/j.cell.2006.10.044. PMID 17190602.
- ↑ Ohno M., Segref A., Bachi A., Wilm M., Mattaj I. W. (2000). «PHAX, a mediator of U snRNA nuclear export whose activity is regulated by phosphorylation». Cell 101 (2): 187—198. DOI:10.1016/S0092-8674(00)80829-6. PMID 10786834.
- ↑ Huber J., Cronshagen U., Kadokura M., Marshallsay C., Wada T., Sekine M., Lührmann R. (1998). «Snurportin1, an m3G-cap-specific nuclear import receptor with a novel domain structure». EMBO J. 17 (14): 4114—4126. DOI:10.1093/emboj/17.14.4114. PMID 9670026.
- ↑ Murthy K. G., Park P., Manley J. L. (1991). «[www.ncbi.nlm.nih.gov/pmc/articles/PMC328187/ A nuclear micrococcal-sensitive, ATP-dependent exoribonuclease degrades uncapped but not capped RNA substrates]». Nucleic Acids Res. 19 (10): 2685—2692. PMID 1710342.
- ↑ Maquat L. E. (2004). «Nonsense-mediated mRNA decay: splicing, translation and mRNP dynamics». Nat. Rev. Mol. Cell Biol. 5 (2): 89—99. DOI:10.1038/nrm1310. PMID 15040442.
- ↑ Jackson R. J., Hellen C. U., Pestova T. V. (2010). «The mechanism of eukaryotic translation initiation and principles of its regulation». Nat. Rev. Mol. Cell Biol. 11 (2): 113—127. DOI:10.1038/nrm2838. PMID 20094052.
- ↑ Sonenberg N., Hinnebusch A. G. (2009). «Regulation of translation initiation in eukaryotes: mechanisms and biological targets». Cell 136 (4): 731—745. DOI:10.1016/j.cell.2009.01.042. PMID 19239892.
- ↑ 1 2 Ling S. H., Qamra R., Song H. (2011). «Structural and functional insights into eukaryotic mRNA decapping». Wiley Interdiscip Rev RNA 2 (2): 193—208. DOI:10.1002/wrna.44. PMID 21957006.
- ↑ 1 2 Garneau N. L., Wilusz J., Wilusz C. J. (2007). «The highways and byways of mRNA decay». Nat. Rev. Mol. Cell Biol. 8 (2): 113—126. DOI:10.1038/nrm2104. PMID 17245413.
- ↑ Otsuka Y., Kedersha N. L., Schoenberg D. R. (2009). «Identification of a cytoplasmic complex that adds a cap onto 5'-monophosphate RNA». Mol. Cell. Biol. 29 (8): 2155—2167. DOI:10.1128/MCB.01325-08. PMID 19223470.
Литература
- Translational Control in Biology and Medicine (2007) Edited by N. Sonenberg, J.W.B. Hershey and M.B. Mathews. 934 pages. Cold Spring Harbor, NY: Cold Spring Harbor Press
- Sonenberg N. (2008) «eIF4E, the mRNA cap-binding protein: from basic discovery to translational research», Biochemistry and Cell Biology 86, 178—183
- Rhoads R.E. (2009) «eIF4E: new family members, new binding partners, new roles», Journal of Biological Chemistry 284, 16711-16715
- Schoenberg D.R. and Maquat L.E. (2009) «Re-capping the message», Trends in Biochemical Sciences 34, 435—442
- Cowling V.H. (2009) «Regulation of mRNA cap methylation», Biochemical Journal 425, 295—302
- Sonenberg N. and Hinnebusch A.G. (2009) «Regulation of translation initiation in eukaryotes: mechanisms and biological targets», Cell 136, 731—745
- Van Der Kelen K., Beyaert R., Inze D. and De Veylder L. (2009) «Translational control of eukaryotic gene expression», Critical Reviews in Biochemistry and Molecular Biology 44,143-168
- Jackson R.J., Hellen C.U.T. and Pestova T.V. (2010) «The mechanism of eukaryotic translation initiation and principles of its regulation», Nature Reviews Molecular Cell Biology 10, 113—127
Ссылки
Категория:- Экспрессия генов
Wikimedia Foundation. 2010.